DNA Sequence Classification
This tutorial demonstrates how to use Milvus, the open-source vector database, to build a DNA sequence classification model.
The ML model and third-party software used include:
- CountVectorizer
- MySQL
- Towhee
DNA sequence is a popular concept in gene traceability, species identification, disease diagnosis, and many more areas. Whereas all industries starve for a more intelligent and efficient research method, artificial intelligence has attracted much attention especially from biological and medical domains. More and more scientists and researchers are contributing to machine learning and deep learning in the field of bioinformatics. To make experimental results more convincing, one common option is to increase sample size. The collaboration with big data in genomics brings more possibilities of application in reality. However, the traditional sequence alignment has limitations, making it unsuitable for large datasets. In order to make less trade-off in reality, vectorization is a good choice for a large dataset of DNA sequences.
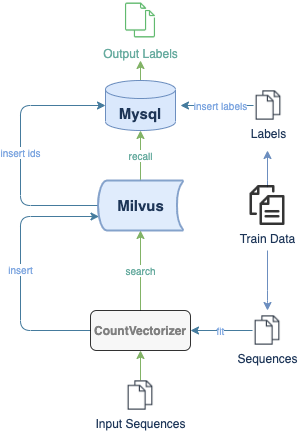
In this tutorial, you will learn how to build a DNA sequence classification model. This tutorial uses CountVectorizer to extract features of DNA sequences and convert them into vectors. Then, these vectors are stored in Milvus and their corresponding DNA classes are stored in MySQL. Users can conduct a vector similarity search in Milvus and recall the corresponding DNA classification from MySQL.
 dna
dna