HDBSCAN Clustering with Milvus
Data can be transformed into embeddings using deep learning models, which capture meaningful representations of the original data. By applying an unsupervised clustering algorithm, we can group similar data points together based on their inherent patterns. HDBSCAN (Hierarchical Density-Based Spatial Clustering of Applications with Noise) is a widely used clustering algorithm that efficiently groups data points by analyzing their density and distance. It is particularly useful for discovering clusters of varying shapes and sizes. In this notebook, we will use HDBSCAN with Milvus, a high-performance vector database, to cluster data points into distinct groups based on their embeddings.
HDBSCAN (Hierarchical Density-Based Spatial Clustering of Applications with Noise) is a clustering algorithm that relies on calculating distances between data points in embedding space. These embeddings, created by deep learning models, represent data in a high-dimensional form. To group similar data points, HDBSCAN determines their proximity and density, but efficiently computing these distances, especially for large datasets, can be challenging.
Milvus, a high-performance vector database, optimizes this process by storing and indexing embeddings, allowing for fast retrieval of similar vectors. When used together, HDBSCAN and Milvus enable efficient clustering of large-scale datasets in embedding space.
In this notebook, we will use the BGE-M3 embedding model to extract embeddings from a news headline dataset, utilize Milvus to efficiently calculate distances between embeddings to aid HDBSCAN in clustering, and then visualize the results for analysis using the UMAP method. This notebook is a Milvus adapation of Dylan Castillo’s article.
Preparation
download news dataset from https://www.kaggle.com/datasets/dylanjcastillo/news-headlines-2024/
$ pip install "pymilvus[model]"
$ pip install hdbscan
$ pip install plotly
$ pip install umap-learn
Download Data
Download news dataset from https://www.kaggle.com/datasets/dylanjcastillo/news-headlines-2024/, extract news_data_dedup.csv and put it into current directory.
Extract Embeddings to Milvus
We will create a collection using Milvus, and extract dense embeddings using BGE-M3 model.
import pandas as pd
from dotenv import load_dotenv
from pymilvus.model.hybrid import BGEM3EmbeddingFunction
from pymilvus import FieldSchema, Collection, connections, CollectionSchema, DataType
load_dotenv()
df = pd.read_csv("news_data_dedup.csv")
docs = [
f"{title}\n{description}" for title, description in zip(df.title, df.description)
]
ef = BGEM3EmbeddingFunction()
embeddings = ef(docs)["dense"]
connections.connect(uri="milvus.db")
- If you only need a local vector database for small scale data or prototyping, setting the uri as a local file, e.g.
./milvus.db, is the most convenient method, as it automatically utilizes Milvus Lite to store all data in this file. - If you have large scale of data, say more than a million vectors, you can set up a more performant Milvus server on Docker or Kubernetes. In this setup, please use the server address and port as your uri, e.g.
http://localhost:19530. If you enable the authentication feature on Milvus, use “: ” as the token, otherwise don’t set the token. - If you use Zilliz Cloud, the fully managed cloud service for Milvus, adjust the
uriandtoken, which correspond to the Public Endpoint and API key in Zilliz Cloud.
fields = [
FieldSchema(
name="id", dtype=DataType.INT64, is_primary=True, auto_id=True
), # Primary ID field
FieldSchema(
name="embedding", dtype=DataType.FLOAT_VECTOR, dim=1024
), # Float vector field (embedding)
FieldSchema(
name="text", dtype=DataType.VARCHAR, max_length=65535
), # Float vector field (embedding)
]
schema = CollectionSchema(fields=fields, description="Embedding collection")
collection = Collection(name="news_data", schema=schema)
for doc, embedding in zip(docs, embeddings):
collection.insert({"text": doc, "embedding": embedding})
print(doc)
index_params = {"index_type": "FLAT", "metric_type": "L2", "params": {}}
collection.create_index(field_name="embedding", index_params=index_params)
collection.flush()
Construct the Distance Matrix for HDBSCAN
HDBSCAN requires calculating distances between points for clustering, which can be computationally intensive. Since distant points have less influence on clustering assignments, we can improve efficiency by calculating the top-k nearest neighbors. In this example, we use the FLAT index, but for large-scale datasets, Milvus supports more advanced indexing methods to accelerate the search process. Firstly, we need to get a iterator to iterate the Milvus collection we previously created.
import hdbscan
import numpy as np
import pandas as pd
import plotly.express as px
from umap import UMAP
from pymilvus import Collection
collection = Collection(name="news_data")
collection.load()
iterator = collection.query_iterator(
batch_size=10, expr="id > 0", output_fields=["id", "embedding"]
)
search_params = {
"metric_type": "L2",
"params": {"nprobe": 10},
} # L2 is Euclidean distance
ids = []
dist = {}
embeddings = []
We will iterate all embeddings in the Milvus collection. For each embedding, we will search its top-k neighbors in the same collection, get their ids and distances. Then we also need to build a dictionary to map original ID to a continuous index in the distance matrix. When finished, we need to create a distance matrix which initialized with all elements as infinity and fill the elements we searched. In this way, the distance between far away points will be ignored. Finally we use HDBSCAN library to cluster the points using the distance matrix we created. We need to set metric to ‘precomputed’ to indicate the data is distance matrix rather than origianl embeddings.
while True:
batch = iterator.next()
batch_ids = [data["id"] for data in batch]
ids.extend(batch_ids)
query_vectors = [data["embedding"] for data in batch]
embeddings.extend(query_vectors)
results = collection.search(
data=query_vectors,
limit=50,
anns_field="embedding",
param=search_params,
output_fields=["id"],
)
for i, batch_id in enumerate(batch_ids):
dist[batch_id] = []
for result in results[i]:
dist[batch_id].append((result.id, result.distance))
if len(batch) == 0:
break
ids2index = {}
for id in dist:
ids2index[id] = len(ids2index)
dist_metric = np.full((len(ids), len(ids)), np.inf, dtype=np.float64)
for id in dist:
for result in dist[id]:
dist_metric[ids2index[id]][ids2index[result[0]]] = result[1]
h = hdbscan.HDBSCAN(min_samples=3, min_cluster_size=3, metric="precomputed")
hdb = h.fit(dist_metric)
After this, the HDBSCAN clustering is finished. We can get some data and show its cluster. Note some data will not be assigned to any cluster, which means they are noise, because they are located at some sparse region.
Clusters Visualization using UMAP
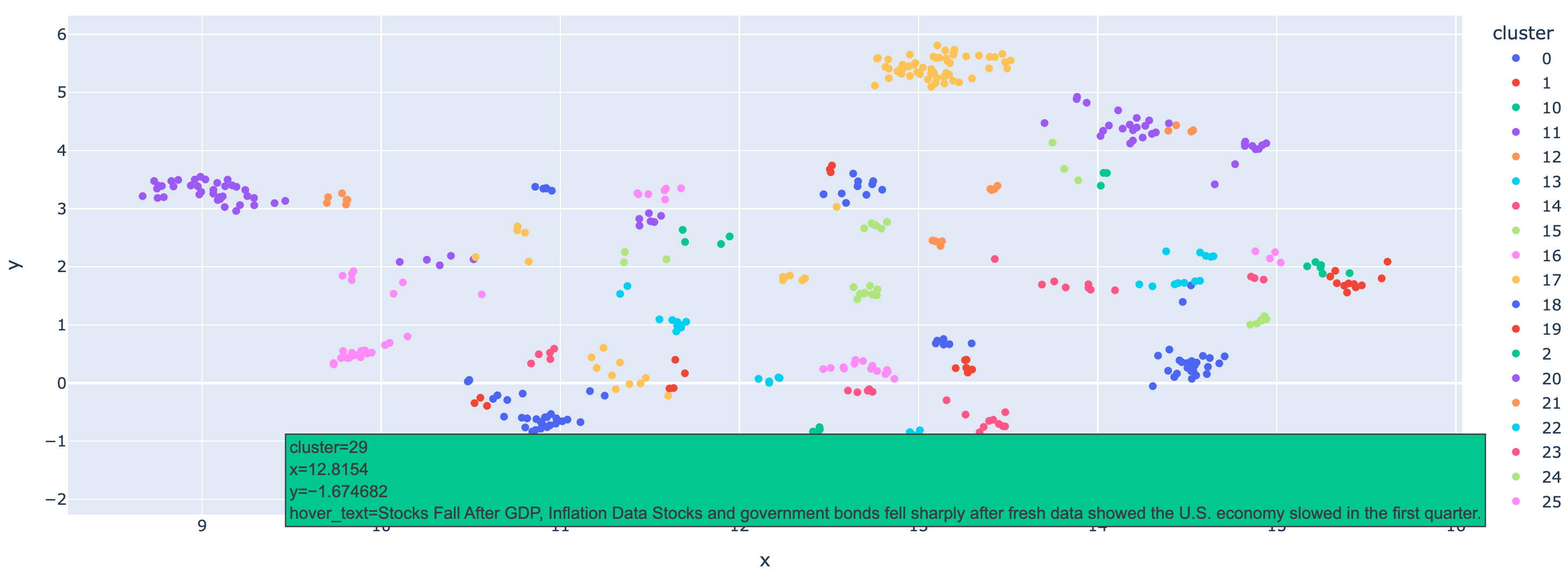
We have already clustered the data using HDBSCAN and can get the labels for each data point. However using some visualization techniques, we can get the whole picture of the clusters for a intuitional analysis. Now we are going the use UMAP to visualize the clusters. UMAP is a efficient methodused for dimensionality reduction, preserving the structure of high-dimensional data while projecting it into a lower-dimensional space for visualization or further analysis. With it, we can visualize original high-dimensional data in 2D or 3D space, and see the clusters clearly. Here again, we iterate the data points and get the id and text for original data, then we use ploty to plot the data points with these metainfo in a figure, and use different colors to represent different clusters.
import plotly.io as pio
pio.renderers.default = "notebook"
umap = UMAP(n_components=2, random_state=42, n_neighbors=80, min_dist=0.1)
df_umap = (
pd.DataFrame(umap.fit_transform(np.array(embeddings)), columns=["x", "y"])
.assign(cluster=lambda df: hdb.labels_.astype(str))
.query('cluster != "-1"')
.sort_values(by="cluster")
)
iterator = collection.query_iterator(
batch_size=10, expr="id > 0", output_fields=["id", "text"]
)
ids = []
texts = []
while True:
batch = iterator.next()
if len(batch) == 0:
break
batch_ids = [data["id"] for data in batch]
batch_texts = [data["text"] for data in batch]
ids.extend(batch_ids)
texts.extend(batch_texts)
show_texts = [texts[i] for i in df_umap.index]
df_umap["hover_text"] = show_texts
fig = px.scatter(
df_umap, x="x", y="y", color="cluster", hover_data={"hover_text": True}
)
fig.show()
 image
image
Here, we demonstrate that the data is well clustered, and you can hover over the points to check the text they represent. With this notebook, we hope you learn how to use HDBSCAN to cluster embeddings with Milvus efficiently, which can also be applied to other types of data. Combined with large language models, this approach allows for deeper analysis of your data at a large scale.
